近些年来,以RNA m6A修饰为代表的RNA 修饰研究非常火热。RNA修饰参与了细胞中的多个关键的生理和病理调控过程,与人类疾病发生发展紧密相关。研究团队前期开发了数据库-m6Avar, 可以让用户挖掘影响m6A修饰的潜在变异,并与各种疾病进行关联。数据库受到了广泛关注和使用。为了让用户能够查阅更多的RNA修饰相关变异位点信息,我们团队对m6AVar进行了重大更新,目前包括9种RNA修饰类型,并且改进了数据库的可视化和注释功能。此外,用户还可以通过UCSC基因组浏览器访问RMVar,可以更加方便的与其他基因组特征进行关联分析。
2020年10月9日,中山大学肿瘤防治中心生物信息平台任间教授,左志向副研究员与附属第一医院谢宇斌副研究员合作,在Nucleic Acids Research (IF:11.501)发表了涉及RNA修饰的功能性变异的更新数据库--RMVar (http://rmvar.renlab.org )。值得一提的是,该研究团队在2018年发布了影响RNA m6A修饰的相关突变数据库M6Avar(http://m6avar.renlab.org ),两年引用达60余次(google scholar)。

研究表明,RNA修饰相关变异与细胞中的许多生物过程失调密切相关,从而导致诸如癌症的严重疾病。涉及功能相关突变,尤其是癌症突变,可以显著改变RNA修饰的状态,导致RNA修饰位点的获得(gain)或丢失(loss)。
RMVar则是专门设计来收集此类功能性变异的RNA数据,旨在为揭示RNA修饰变异体潜在的功能提供帮助。
数据库首页
目前,RMVar收集了941,955个修饰位点以及1,678,126个RNA修饰相关突变的数据,其中包括来自于dbSNP 和HGVD的1,457,898个RNA修饰相关的种系突变数据,以及220,228个来自于TCGA, ICGC 和COSMIC的体细胞突变数据
,涵盖了9种最常见的RNA修饰,包括N6-methyladenosine (m6A), N6-dimethyladenosine (m6Am), N1-methyladenosine (m1A), pseudouridine (ψ), 5-methylcytosine (m5C), ribose methylations (2′-O-Me), 7-Methylguanosine (m7G), 5-methyluridine (m5U) 和Adenosine-to-inosine (A-to-I)。
检索模块:
为了方便使用,RMVar提供了多种用户友好的Web界面及接口。
1) Quick Search
首先,在首页我们提供了简单快速查询功能,用户可以通过选择RMVar_ID、SNP的RsID、基因名,染色体的特定区域或者疾病名等进行快速搜索。
Quick Search界面
2) Advance Search
另外,RMVar还提供了一个Advance Search页面及接口,用户可以自定义组合多个条件来获取其更加精确的查询结果。
Advance Search界面
3) 检索结果展示
对于检索结果,RMVar提供了一个相关条目的总览表以及检索到的数据的统计图。
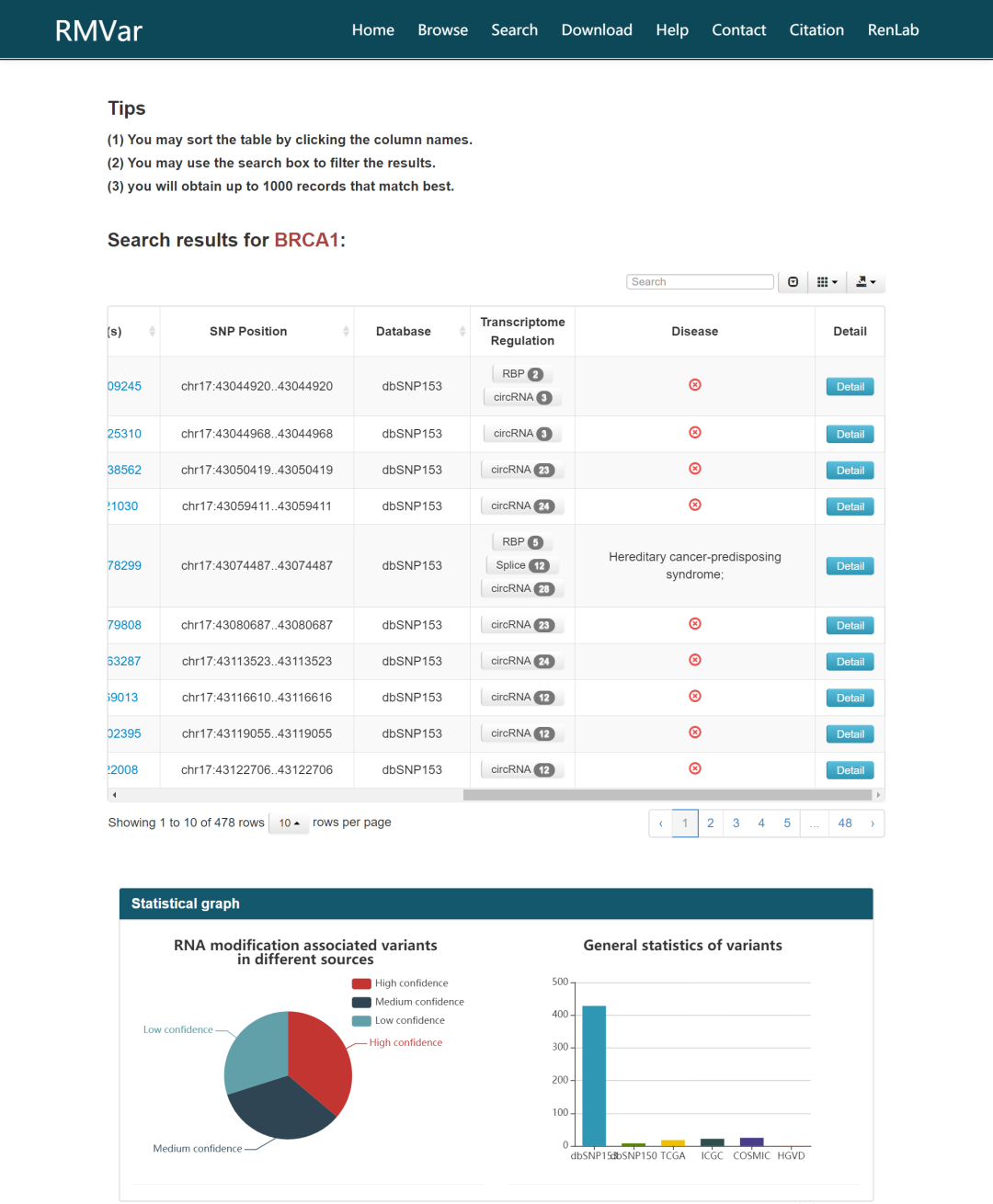
检索结果页面
点击最右边的“Detail”按钮将链接到该数据条目的详细信息页面,或者点击“Transcriptome Regulation”列的相关按钮直接链接到详细信息页面的相关信息所在部分。
此外,RMVar还嵌入了基因组浏览器,以可视化分析结果。用户点击第一个基本信息表格最下方的“Visualize in genome browser”按钮则可以查看其可视化效果,在该页面将展示与该记录相关的转录本注释信息,包括相关的peak和reads覆盖度信息等。另外,用户可以自由缩放并且自定义选择需要展示的轨道等,以此来查看特定区域或者特定注释轨道的信息。
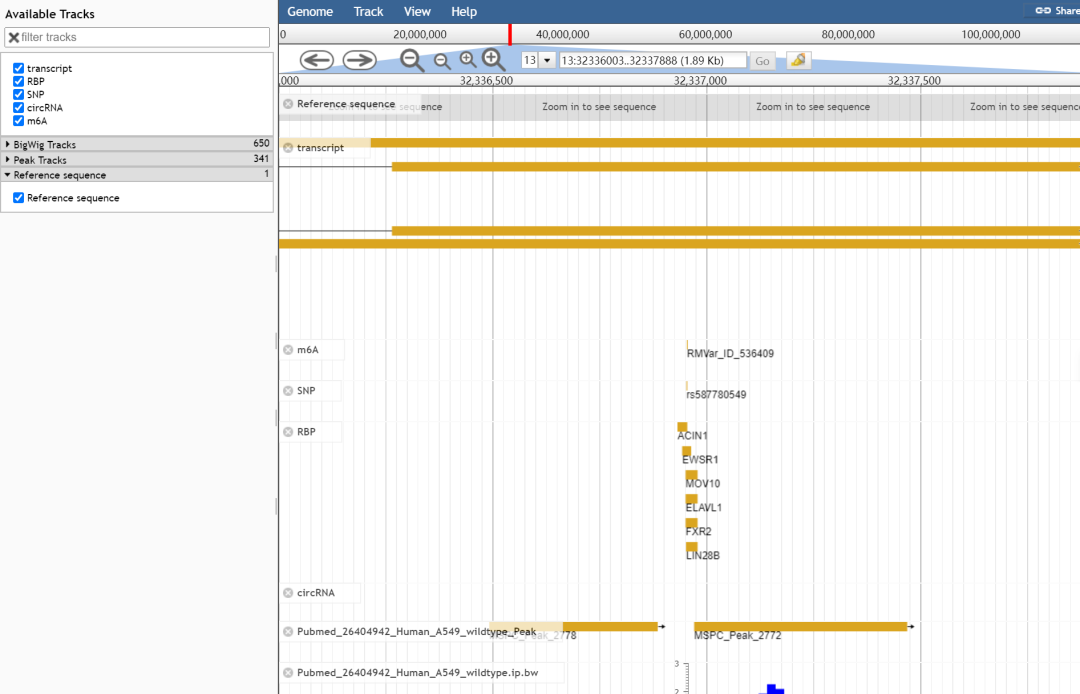
基因组浏览器页面
Browse模块:
在Browse页面,用户可以首先选择RNA修饰类型,接下来可以通过选择Germline来查询不同物种的种系变异数据,或者选择Somatic来查询人类不同癌症或者组织的体细胞变异数据。之后在该页面将以多种可视化图表的形式展示其所选条件下的RNA数据的统计信息以及相关条目信息。
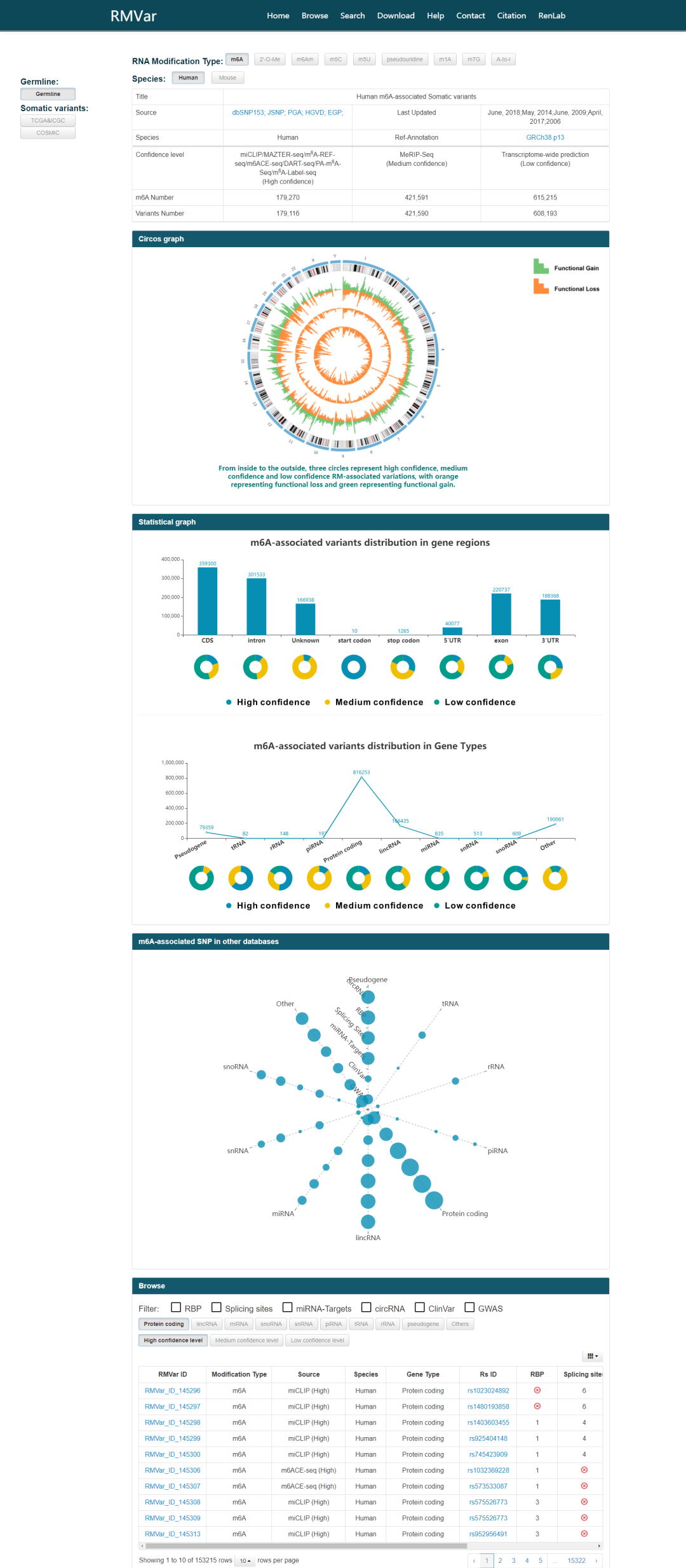
图9. Browse页面条件筛选部分
最后对于查询到的具体数据条目,用户可以通过RNA修饰涉及的相关功能(RBP, Splicing sites, miRNA-Targets, circRNA, ClinVar, GWAS等),Gene type或者confidence level来进一步筛选,并且可以通过点击表格里的RMVar_ID列来链接到该数据的详细信息页面。
Download模块:
最后数据库通过详细的分类信息提供了所有数据下载,方便用户进一步的分析与挖掘。
图12. 数据库Download页面
在UCSC上查看RMVar track:
戳重点: 应UCSC邀请,我们在UCSC的Genome browser上提供了RMvar的track,方便和其他层面的数据进行关联分析和对比。如下图分别为小鼠和人的部分RMvar位点展示
小鼠
http://genome.ucsc.edu/cgi-bin/hgTracks?db=mm10&hubUrl=http://rmvar.renlab.org/RMVarhub/hub.txt
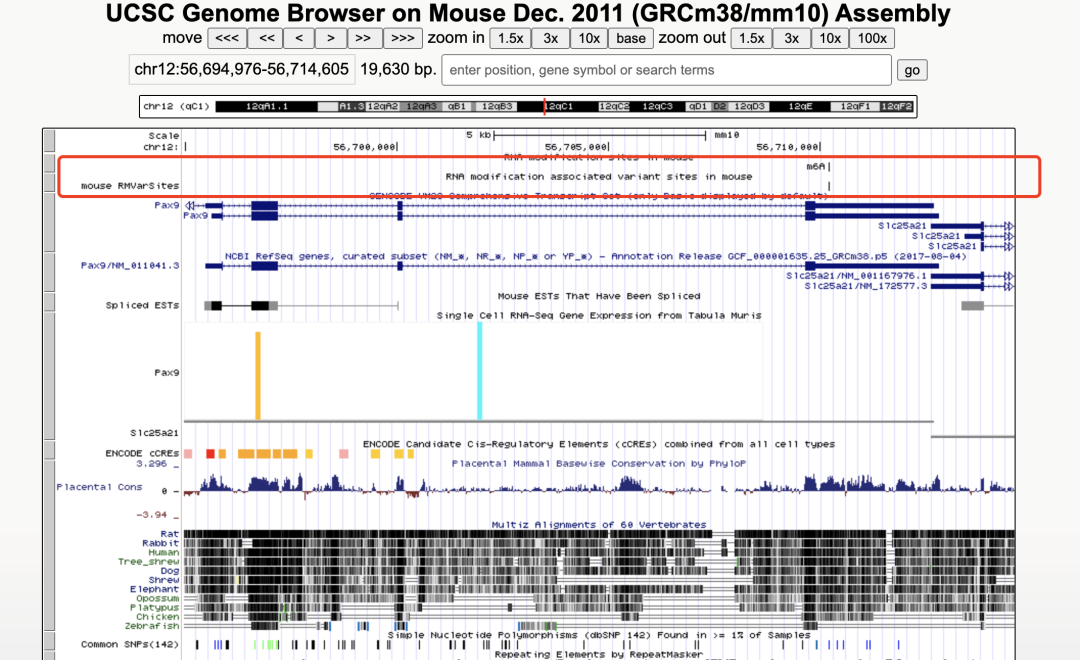
图13. UCSC中genome browser的RMvar小鼠位点界面展示
人
http://genome.ucsc.edu/cgi-bin/hgTracks?db=hg38&hubUrl=http://rmvar.renlab.org/RMVarhub/hub.txt

图14. UCSC中Genome browser的RMvar人RNA修饰位点track展示














